近日,农林科学国际期刊《Industrial Crops & Products》在线发表了bat365在线平台网站棉花创新团队的研究论文“Unraveling the genetic basis of superior traits in Gossypium barbadense: From phenotype to genotype”。该研究利用海岛棉RIL群体,通过高密度的遗传连锁图谱结合5个环境的表型数据定位到了169个QTL,发现了两个与衣分紧密连锁的bin标记可作为育种标记。此外,结合RNA-seq,揭示了棉纤维不同发育阶段基因表达的动态变化,构建了纤维次生细胞壁发育的基因调控网络,挖掘到了8个调控纤维品质性状相关的候选基因,阐明了我国海岛棉育种史上优良位点qFL_D04_1的遗传基础。

棉花是世界上广泛种植的纤维作物,针对其纤维品质的改良一直是研究热点,创制和选育出具有高产量和优异纤维品质的种质资源也成为棉花育种工作者长期努力的方向与目标。但对棉花的研究主要集中在陆地棉,海岛棉的研究进展相对缓慢。对重要性状的遗传位点和转录调控机制的了解有限,优良性状发育的遗传基础仍不清楚。随着海岛棉(3-79、海7124和Pima90)的基因组被测序,为研究海岛棉的遗传学和基因组学提供了重要的基因组资源,加快了利用标记辅助选择(MAS)或全基因组选择(GS)育种的进程。从分子水平的机理研究有助于更好地理解海岛棉优良性状的调控机制,因此,有必要针对海岛棉优良性状在不同环境中均稳定表达的主效QTL开展定位及分子机制解析工作,并通过开发紧密连锁的分子标记或者功能标记,借助于标记辅助选择和转基因育种手段来快速改良棉花的产量和品质。
随着测序技术的进步和成本的不断降低,导致全基因组范围内标记开发成本降低且速度加快,由此出现了许多基因定位的方法。比如全基因组关联分析,高密度遗传连锁图谱QTL定位,BSA-seq等。在实际研究中,往往会将多种方法联合起来,例如使用QTL定位或者BSA-seq配合RNA-Seq够对目标性状特异基因快速的完成定位和功能分析。本研究以来自美国的06E2062和新疆大面积推广材料新海21为亲本构建了重组自交系(RIL)群体。综合分析了双亲本和RIL群体的表型特征,以及RIL群体的表型相关性、遗传力和遗传模型。采用全基因组重测序技术构建了包含5295个bin标记,总遗传图谱距离为2721.79cM的高密度遗传连锁图谱。利用5种不同环境下的表型数据检测到169个QTL位点。包含30个与农艺性状相关的QTL,61与产量性状相关QTL个,78个与纤维品质性状相关QTL,发现了两个与衣分紧密连锁的bin标记可作为育种标记。(图1)。
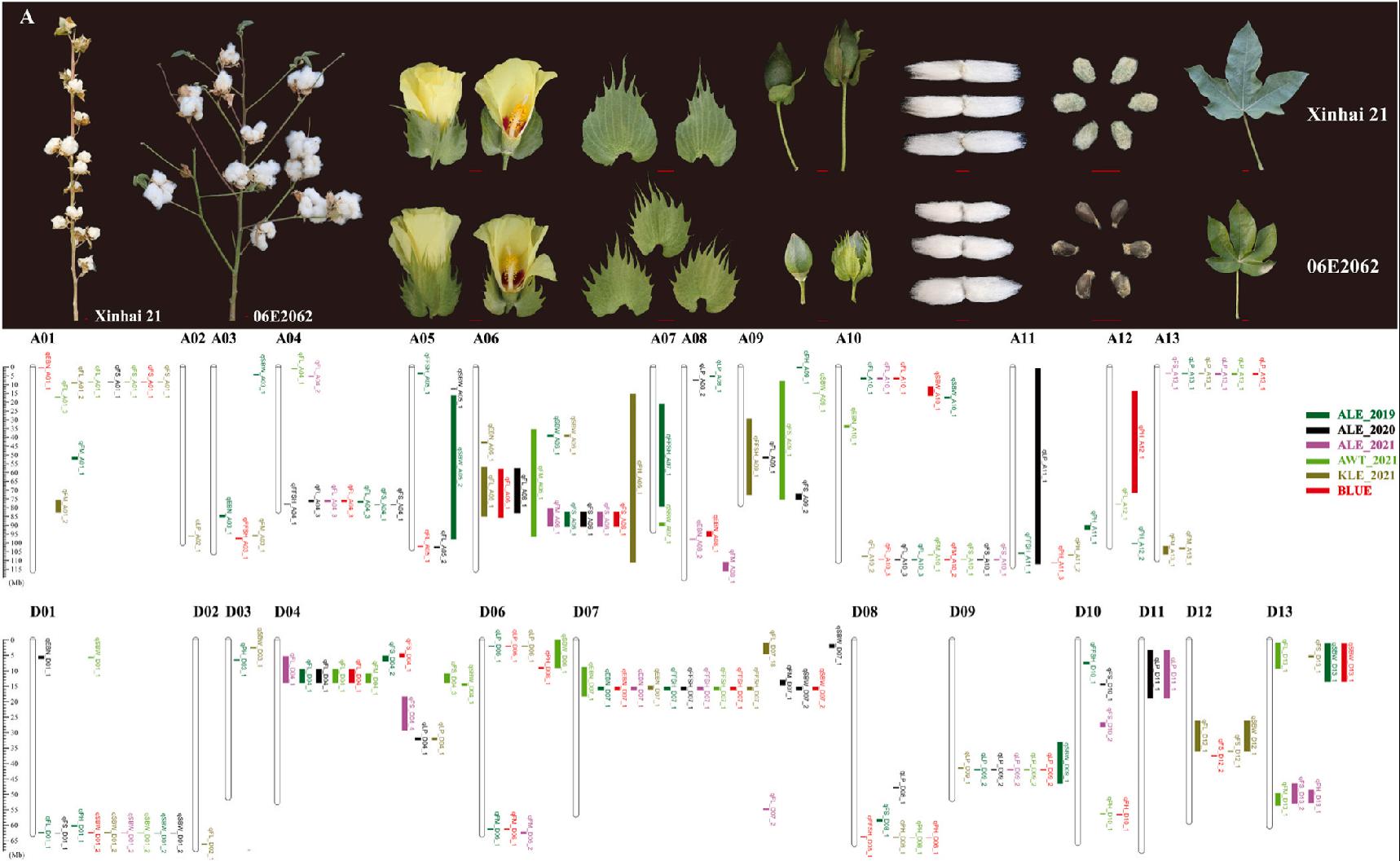
图1 新海21和06E2062的表型特征及8个不同性状的QTL定位
利用RIL群体亲本棉纤维发育在纤维发育的7个时间点的RNA-seq数据,揭示了棉纤维不同发育阶段基因表达的动态变化,并鉴定了纤维次生细胞壁发育和代谢网络中重要的差异表达基因。QTL定位和转录组的联合分析,预测了8个参与调节纤维品质性状的候选基因,我国海岛棉育种史上优良位点qFL_D04_1的遗传基础。本研究为棉花品种改良提供了新的基因资源和潜在育种标记,为了解海岛棉重要性状的遗传基础提供了有价值的理论信息,有效推动了棉花生物技术育种的发展。

图2 海岛棉次生细胞壁发育过程中候选基因的鉴定与分析
bat365在线平台网站棉花创新团队带头人陈全家教授和郑凯副教授为该论文的通讯作者,博士生蔡永生为该论文的第一作者。该研究得到了国家自然科学基金(32301867)、新疆“天山人才培养计划”(2023TSYCLJ0012)、国家育种联合研究(2022MH-01)和新疆维吾尔自治区“揭榜挂帅”(GMLM2023–09)项目的资助。







