沙门菌是世界上主要的食源性病原菌之一,由污染的食品导致消费者产生严重的致病率和致死率。但是,在中国对食品产业链中污染的沙门菌很少采用全基因组测序进行溯源研究。因此,姚刚、刘英玉团队和浙江大学乐敏教授合作应用全基因组测序对中国新疆地区动物食品链中沙门菌的流行病学和溯源研究。
2021年4月在Food Research International(IF 6.475, 1区)期刊杂志上发表了论文“Prevalence and Genomic Investigation of Salmonella Isolates Recovered from Animal Food-chain in Xinjiang, China”。这是我们首次报道了在新疆地区动物源性食品产业链中污染流行的多重耐药沙门菌,及其携带编码的关键毒力因子情况,通过采用全基因组测序平台对沙门菌进行溯源研究有助于对食品产业链上出现的多重耐药菌的鉴定和早期控制。
我们从新疆地区牛羊养殖场、屠宰过程和市场环境中采集了2408份样品进行沙门菌的分离鉴定,分离出314株沙门菌(分离率13.04%)。如图1A通过比较动物源性食品产业链的采样地点,发现养殖场的分离率最高为7.52%(65/864),其次是市场的分离率较高为8.26%(40/484),屠宰场的分离率为7.52%(65/864)。如图1B比较不同动物源样品发现沙门菌在羊源样品中分离率最高为16.58%(157/947),其次是牛源样品中分离率较高为11.22%(143/1275),其他来源样品中分离率较低为7.53%(14/186)。根据卡方检验的统计分析表明,沙门菌分离株的流行率无明显的食品产业链和样品来源的差异。
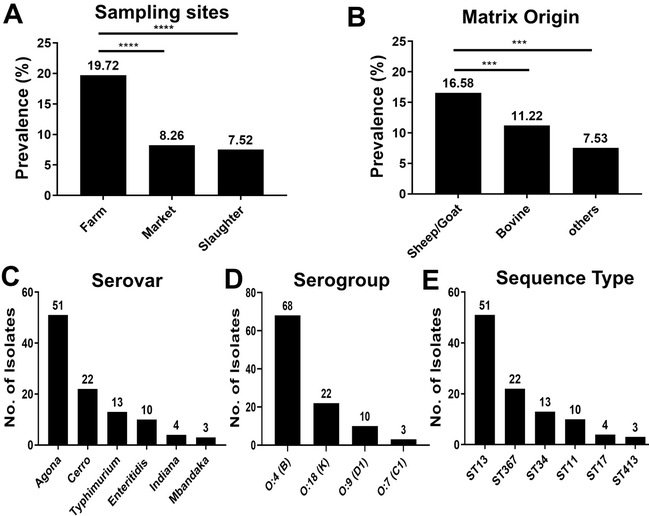
根据不同采样地点和动物种类选择了经鉴定后的103株沙门菌,采用基因组框架图测序方法进行沙门菌的血清型、抗性基因、毒力基因、质粒类型、MLST型等研究。通过全基因组测序分析发现阿贡纳沙门菌为优势血清型(49.51%;51/103),O:4(B)为优势血清组(66.02%;68/103)(如图1C和1D);沙门菌可分为六种ST型(如图1E),每个血清型都有对应的MLST型,主要的基因分型是ST13。主要质粒类型是Col (MGD2)_1和Col (Ye4449)_1,肠炎沙门菌均编码了IncFII (S)_1和IncFIB (S)_1质粒,而且携带有毒力基因;印第安纳沙门菌和肠炎沙门菌编码伤寒毒素的cdtB基因,编码纤毛的spv和pef基因。
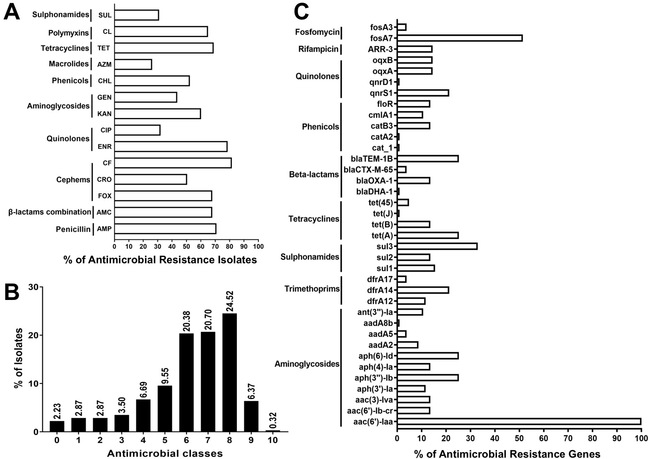
采用肉汤稀释法对314株沙门菌进行了14种抗菌药物的耐药性调查,发现92.04%的沙门菌是多重耐药菌株(如图2B),其中四环素的耐药率是78.03%(245/314),阿莫西林/克拉维酸的耐药率是75.80%(238/314),氨苄青霉素的耐药率是70.70%(222/314)。采用浙江大学建立的Galaxy平台,基于九类抗生素耐药性的37个耐药基因进行预测,发现所有菌株均携带有氨基糖苷的aac(6’)-Iaa基因,有53株(51.45%)沙门菌携带有耐磷霉素的fosA7基因,有34株(33.01%)沙门菌携带有耐磺胺类的sul3基因,有26株(25.24%)沙门菌携带有耐氨基糖苷类的aph(6)-Id基因、四环素类的tet(A)基因和β-内酰胺类的blaTEM-1B基因(图2C)。另一方面,比较分析了表型检测和基因预测的抗生素耐药性数据的差异(图2A和C)。
bat365在线平台网站刘英玉副教授和在读研究生蒋金豆为该文的共同第一作者,姚刚教授和乐敏教授为该文的共同通讯作者,已毕业研究生朱梦含和夏利宁教授参与本研究工作。该研究受到国家十三五国家重点研发计划项目(2018YFD0500504)的支持。
文章链接:https://www.sciencedirect.com/science/article/abs/pii/S0963996921000971







